R 은 통계분석을 위한 아주 유용한 Open source 프로그램이다. 배우기도 용이하고 무엇보다 Open source 프로그램 이기에 비용 부담도 없다는 것이다. 제조기업 현장에서 통계분석이 필요한 여러가지 상황에서 사용할 수 있는 방법을 제시하고자 한다.
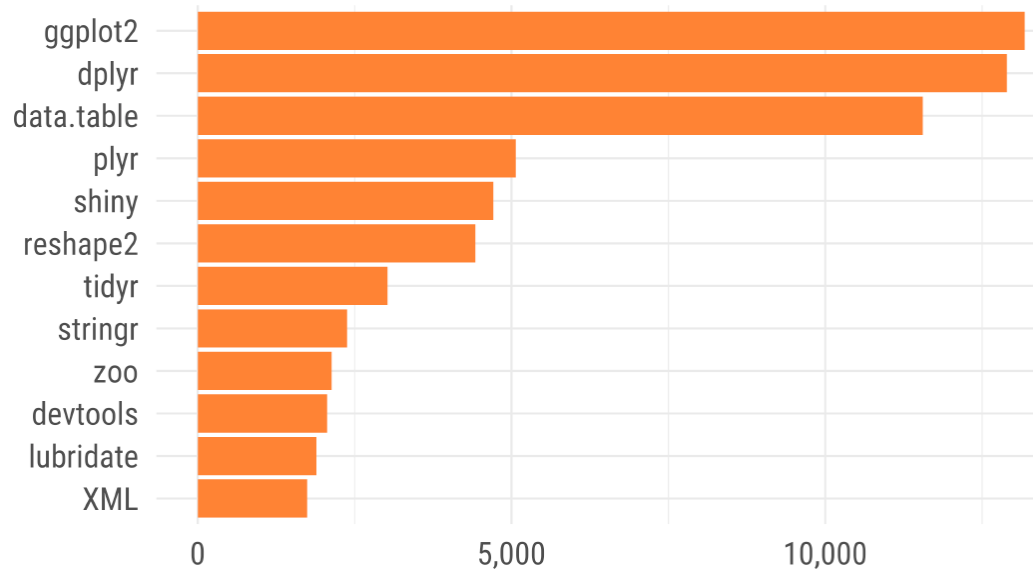
image source : https://statkclee.github.io/data-science/data-science-library.html

image source : https://statkclee.github.io/data-science/data-science-library.html
code 참조 : github